🚀 SMILES 基状态空间编解码器 (SMI - SSED) - MoLMamba
本项目提供了与我们的论文 “A Mamba - Based Foundation Model for Chemistry” 相关的 PyTorch 源代码。该项目基于 Mamba 架构构建了一个化学基础模型,可用于解决化学领域的多种复杂任务,具有重要的科研和应用价值。
🚀 快速开始
预训练模型和训练日志
我们提供了在从 PubChem 精心挑选的约 9100 万个分子数据集上预训练的 SMI - SSED 模型的检查点。该预训练模型在 MoleculeNet 的分类和回归基准测试中表现出色。
根据需求,将 SMI - SSED 的 预训练权重.pt 文件添加到 inference/ 或 finetune/ 目录中。目录结构应如下所示:
inference/
├── smi_ssed
│ ├── smi_ssed.pt
│ ├── bert_vocab_curated.txt
│ └── load.py
和/或:
finetune/
├── smi_ssed
│ ├── smi_ssed.pt
│ ├── bert_vocab_curated.txt
│ └── load.py
复制 Conda 环境
按照以下步骤复制我们的 Conda 环境并安装必要的库:
创建并激活 Conda 环境
conda create --name smi-ssed-env python=3.10
conda activate smi-ssed-env
使用 Conda 安装包
conda install pytorch=2.1.0 pytorch-cuda=11.8 -c pytorch -c nvidia
使用 Pip 安装包
pip install -r requirements.txt
✨ 主要特性
- 多任务支持:支持量子属性预测等多种复杂任务。
- 双格式模型权重:提供 PyTorch (
.pt) 和 safetensors (.bin) 两种格式的模型权重。
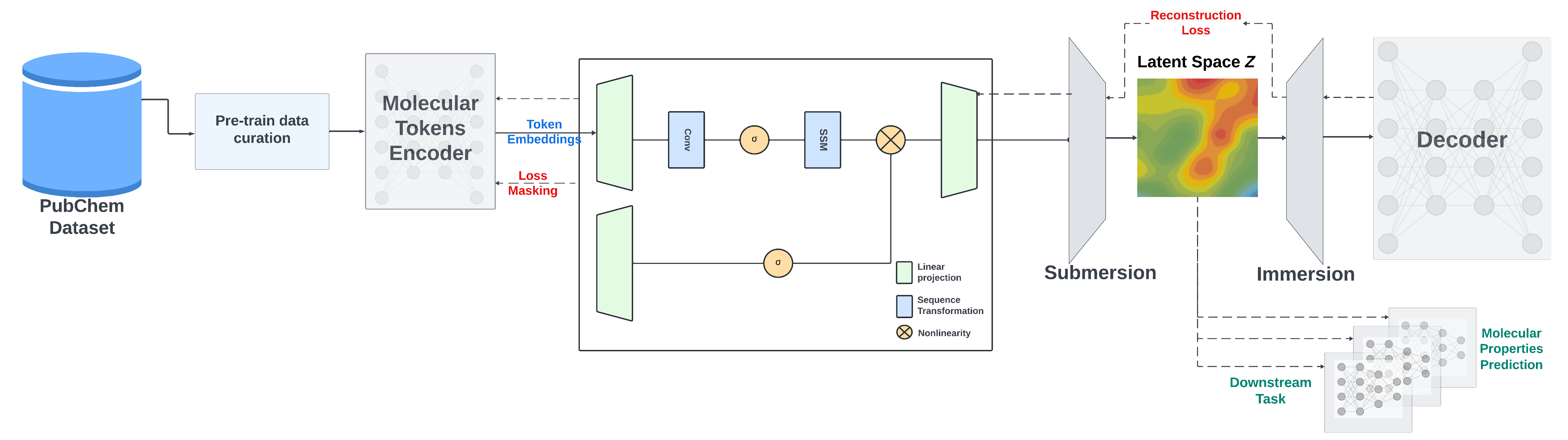
- 预训练策略多样:采用掩码语言模型方法训练编码器部分,以及编码器 - 解码器策略优化 SMILES 重建和改善生成的潜在空间。
📦 安装指南
预训练
对于预训练,我们采用两种策略:使用掩码语言模型方法训练编码器部分,以及使用编码器 - 解码器策略优化 SMILES 重建并改善生成的潜在空间。
SMI - SSED 在来自 PubChem 的 9100 万个经过规范化和精心挑选的 SMILES 上进行预训练,并遵循以下约束条件:
- 预处理期间,化合物过滤为最大长度 202 个标记。
- 编码器训练使用 95/5/0 分割,其中 5% 的数据用于解码器预训练。
- 还使用 100/0/0 分割直接训练编码器和解码器,以提高模型性能。
预训练代码提供了在较小数据集上进行数据处理和模型训练的示例,需要 8 个 A100 GPU。
要预训练 SMI - SSED 模型,请运行:
bash training/run_model_training.sh
使用 train_model_D.py 仅训练解码器,或使用 train_model_ED.py 训练编码器和解码器。
微调
微调数据集和环境可在 finetune 目录中找到。设置好环境后,可以运行以下命令进行微调任务:
bash finetune/smi_ssed/esol/run_finetune_esol.sh
微调训练/检查点资源将在名为 checkpoint_<measure_name> 的目录中可用。
💻 使用示例
基础用法
model = load_smi_ssed(
folder='../inference/smi_ssed',
ckpt_filename='smi_ssed.pt'
)
高级用法
编码 SMILES 为嵌入向量
with torch.no_grad():
encoded_embeddings = model.encode(df['SMILES'], return_torch=True)
解码嵌入向量为 SMILES 字符串
with torch.no_grad():
decoded_smiles = model.decode(encoded_embeddings)
📚 详细文档
特征提取
示例笔记本 smi_ssed_encoder_decoder_example.ipynb 包含加载检查点文件并使用预训练模型进行编码器和解码器任务的代码。它还包括分类和回归任务的示例。模型权重可在 HuggingFace 链接 获取。
📄 许可证
本项目采用 Apache - 2.0 许可证。
其他信息
论文链接
论文 NeurIPS AI4Mat 2024:Arxiv 链接
联系方式
如需更多信息,请联系:eduardo.soares@ibm.com 或 evital@br.ibm.com。
模型图示
模型信息表格
| 属性 |
详情 |
| 模型类型 |
基于 Mamba 的编码器 - 解码器化学基础模型 |
| 训练数据 |
来自 PubChem 的 9100 万个经过规范化和精心挑选的 SMILES |
| 评估指标 |
准确率 |
| 任务类型 |
特征提取 |
| 标签 |
化学、基础模型、AI4Science、材料、分子、safetensors、pytorch、transformer、diffusers |
| 库名称 |
transformers |
 Transformers 支持多种语言
Transformers 支持多种语言 Transformers 支持多种语言
Transformers 支持多种语言 Transformers 英语
Transformers 英语 Transformers 英语
Transformers 英语