模型简介
模型特点
模型能力
使用案例
🚀 Prov - GigaPath
Prov - GigaPath是一个用于数字病理学的全切片基础模型,它基于真实世界的数据构建,为数字病理学的研究提供了有力支持。
🚀 快速开始
安装
在启用了CUDA工具包的NVIDIA A100 Tensor Core GPU机器上进行以下操作:
- 下载我们的代码仓库并进入Prov - GigaPath目录
git clone https://github.com/prov-gigapath/prov-gigapath
cd prov-gigapath
- 安装GigaPath及其依赖项
conda env create -f environment.yaml
conda activate gigapath
pip install -e .
模型下载
可以从HuggingFace Hub访问Prov - GigaPath模型。 你需要同意相关条款才能访问这些模型。获得必要的访问权限后,将你的HuggingFace只读令牌设置为环境变量:
export HF_TOKEN=<huggingface read-only token>
如果你未设置令牌,可能会遇到以下错误:
ValueError: We have no connection or you passed local_files_only, so force_download is not an accepted option.
✨ 主要特性
- 基于真实世界数据构建,为数字病理学提供全切片基础模型。
- 可用于图像特征提取,在数字病理学研究中有广泛应用。
📦 安装指南
在启用了CUDA工具包的NVIDIA A100 Tensor Core GPU机器上:
- 下载仓库:
git clone https://github.com/prov-gigapath/prov-gigapath
cd prov-gigapath
- 安装依赖:
conda env create -f environment.yaml
conda activate gigapath
pip install -e .
💻 使用示例
基础用法
推理 - 切片编码器
首先,加载GigaPath切片编码器:
import timm
from PIL import Image
from torchvision import transforms
import torch
tile_encoder = timm.create_model("hf_hub:prov-gigapath/prov-gigapath", pretrained=True)
transform = transforms.Compose(
[
transforms.Resize(256, interpolation=transforms.InterpolationMode.BICUBIC),
transforms.CenterCrop(224),
transforms.ToTensor(),
transforms.Normalize(mean=(0.485, 0.456, 0.406), std=(0.229, 0.224, 0.225)),
]
)
运行推理以提取切片级特征:
img_path = "images/prov_normal_000_1.png"
sample_input = transform(Image.open(img_path).convert("RGB")).unsqueeze(0)
tile_encoder.eval()
with torch.no_grad():
output = tile_encoder(sample_input).squeeze()
推理 - 幻灯片编码器
要使用幻灯片编码器进行推理,我们需要切片嵌入及其坐标作为输入。首先,加载GigaPath幻灯片编码器:
import gigapath
slide_encoder = gigapath.slide_encoder.create_model("hf_hub:prov-gigapath/prov-gigapath", "gigapath_slide_enc12l768d", 1536)
运行推理以获取幻灯片级嵌入:
slide_encoder.eval()
with torch.no_grad():
output = slide_encoder(tile_embed, coordinates).squeeze()
高级用法
微调 - 使用PCam数据集进行切片级线性探测示例
为方便起见,我们提供了PCam数据集的预提取嵌入。你可以从以下链接下载,注意文件大小为2GB。
wget -nc https://hanoverprod.z21.web.core.windows.net/gigapath/GigaPath_PCam_embeddings.zip -P data/
无需解压此文件。 要运行微调实验,请执行以下脚本:
bash scripts/run_pcam.sh data/GigaPath_PCam_embeddings.zip
微调 - 使用PANDA数据集进行幻灯片级微调示例
为方便起见,我们提供了PANDA数据集的预提取嵌入。你可以从以下链接下载,注意文件大小为32GB,请解压此文件。
wget -nc https://hanoverprod.z21.web.core.windows.net/gigapath/GigaPath_PANDA_embeddings.zip -P data/
unzip -n data/GigaPath_PANDA_embeddings.zip -d data/
要运行微调实验,请执行以下脚本:
bash scripts/run_panda.sh data/GigaPath_PANDA_embeddings/h5_files
📚 详细文档
模型概述
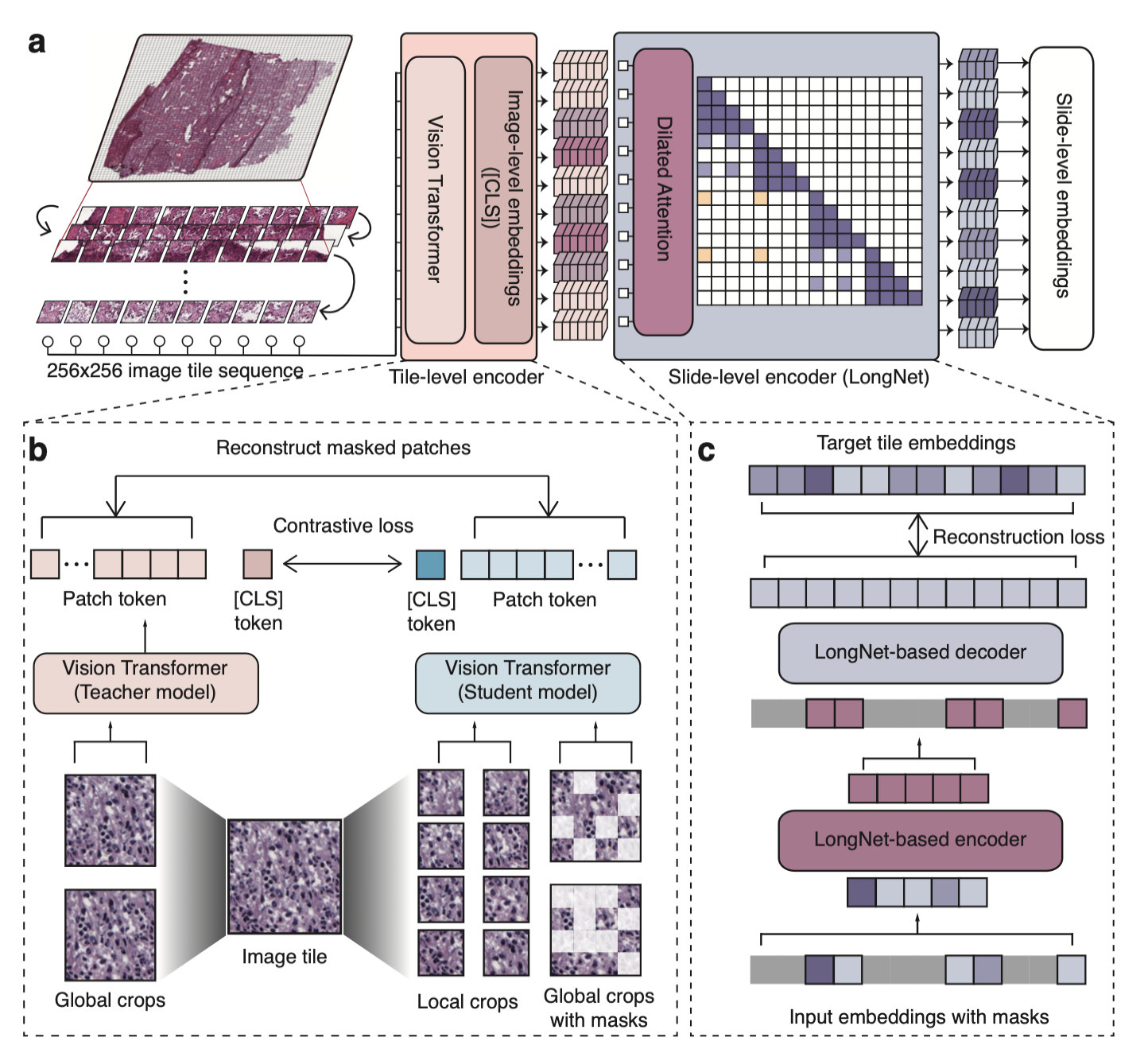
Prov - GigaPath模型架构概述
模型用途
预期用途
数据、代码和模型检查点仅用于(I)未来病理学基础模型的研究和(II)参考论文中实验结果的重现。数据、代码和模型检查点不用于临床护理或任何临床决策目的。
主要预期用途
主要预期用途是支持AI研究人员重现和扩展这项工作。GigaPath有助于探索数字病理切片数据的预训练和编码。
超出范围的用途
模型的任何部署用例(商业或其他)都超出了范围。尽管我们使用了广泛的公开研究基准对模型进行了评估,但模型和评估仅用于研究目的,不用于部署用例。
使用和许可声明
该模型并非作为医疗设备、临床支持、诊断工具或其他用于疾病诊断、治疗、缓解、治疗或预防的技术提供临床使用。该模型并非设计或旨在替代专业医疗建议、诊断、治疗或判断,不应如此使用。所有用户有责任审查开发模型的输出,以确定模型是否满足用户需求,并在任何临床使用前验证和评估模型。
📄 许可证
致谢
我们感谢构建此项目所依赖的优秀仓库的作者和开发者:DINOv2、MAE、Timm和TorchScale。他们的贡献对我们的工作非常宝贵。
引用
如果您发现Prov - GigaPath对您的研究和应用有用,请使用以下BibTeX进行引用:
@article{xu2024gigapath,
title={A whole-slide foundation model for digital pathology from real-world data},
author={Xu, Hanwen and Usuyama, Naoto and Bagga, Jaspreet and Zhang, Sheng and Rao, Rajesh and Naumann, Tristan and Wong, Cliff and Gero, Zelalem and González, Javier and Gu, Yu and Xu, Yanbo and Wei, Mu and Wang, Wenhui and Ma, Shuming and Wei, Furu and Yang, Jianwei and Li, Chunyuan and Gao, Jianfeng and Rosemon, Jaylen and Bower, Tucker and Lee, Soohee and Weerasinghe, Roshanthi and Wright, Bill J. and Robicsek, Ari and Piening, Brian and Bifulco, Carlo and Wang, Sheng and Poon, Hoifung},
journal={Nature},
year={2024},
publisher={Nature Publishing Group UK London}
}
样本数据下载
可以从以下链接访问Prov - Path数据的样本去标识子集 [1, 2]。
项目链接
作者信息
Hanwen Xu*, Naoto Usuyama*, Jaspreet Bagga, Sheng Zhang, Rajesh Rao, Tristan Naumann, Cliff Wong, Zelalem Gero, Javier González, Yu Gu, Yanbo Xu, Mu Wei, Wenhui Wang, Shuming Ma, Furu Wei, Jianwei Yang, Chunyuan Li, Jianfeng Gao, Jaylen Rosemon, Tucker Bower, Soohee Lee, Roshanthi Weerasinghe, Bill J. Wright, Ari Robicsek, Brian Piening, Carlo Bifulco, Sheng Wang, Hoifung Poon (*同等贡献)
 Transformers
Transformers Transformers
Transformers Transformers
Transformers Transformers
Transformers Transformers
Transformers Transformers
Transformers Transformers
Transformers Transformers
Transformers Transformers 支持多种语言
Transformers 支持多种语言 Transformers 英语
Transformers 英语